Résumés
Résumé
La technique PRINS (primed in situ labelling ou synthèse in situ amorcée) est une technique non isotopique de cytogénétique moléculaire qui permet l’analyse microscopique de séquences d’ADN nucléaire sur chromosomes mitotiques et dans les noyaux interphasiques. Elle constitue une excellente alternative à la technique FISH (fluorescence in situ hybridization). Parmi les multiples avantages qui caractérisent la technique PRINS, on retiendra sa grande spécificité, sa sensibilité, sa rapidité d’exécution et son faible coût de revient. La réaction PRINS peut être combinée à d’autres techniques telles que la technique FISH, l’immunofluorescence indirecte et la technique de « translation de coupure ». Les développements récents témoignent du grand potentiel de cette technique qui permet notamment la détection de gènes en copie unique impliqués dans certaines maladies. La variété de ces applications font de la technique PRINS un outil cytogénétique de choix, en clinique comme en recherche.
Summary
As a non-isotopic molecular cytogenetic technique, the primed insitu (PRINS) labelling reaction represents a major technological progress achieved in the past decade. It has become a routine technique for the microscopic visualization of specific DNA sequences in cells and nuclei and constitutes a good alternative to the fluorescence in situ hybridization (FISH) procedure. Among the multiple advantages that characterize the PRINS technique, specificity, rapidity, reliability, reproducibility, and cost-effectiveness can be mentioned. PRINS can be in addition associated with other techniques like FISH, indirect immunofluorescence, and nick translation. The most recent developments show the great potential of this technique. Now PRINS can be used to study single-copy genes and, consequently, can be routinely used to investigate deletions associated with microdeletion syndromes. Therefore, the PRINS technique has the potential to become a widely used molecular cytogenetic tool in clinics and research. This short review presents how the PRINS technique contributed to further the understanding of biological phenomena and describes the different possibilities and applications of the PRINS method in several biological and clinical fields (pre-implantation testing, prenatal, constitutional and oncologic genetic diagnosis).
Corps de l’article
Nous assistons, depuis l’avènement du marquage en bandes des chromosomes, à une véritable révolution technologique en cytogénétique humaine et animale. Après l’introduction en 1969 par M.L. Pardue et J.G. Gall [1] de l’hybridation in situ isotopique, il est devenu possible d’analyser des séquences d’ADN à l’intérieur des cellules sans altérer leur morphologie. En 1986, Pinkel et al. [2] puis Cremer et al. [3] ont introduit l’hybridation in situ fluorescente ou FISH (fluorescence in situ hybridization) avec des sondes non radioactives combinant les principes d’hybridation de l’ADN aux techniques d’immunofluorescence. En 1989, la technique de marquage non radioactif par synthèse in situ amorcée ou PRINS (primed in situ labelling) décrite par Koch et al. [4] va alors représenter un outil de recherche et de diagnostic en cytogénétique moléculaire (Figure 1). Comparée à la technique FISH, cette méthode offre de multiples avantages, notamment la simplicité de son protocole, la spécificité du marquage obtenu et son faible coût (Tableau I).
Figure 1
Schéma illustrant les deux étapes de la technique PRINS (marquage non isotopique par synthèse in situ amorcée) et ses principales applications.
Un volume d’environ 45 μl d’un mélange réactionnel constitué entre autre d’une amorce (courte séquence oligonucléotidique de 18 à 35 nucléotides), de trois nucléotides non marqués et d’un nucléotide marqué (soit avec un haptène non fluorescent comme la biotine ou la digoxigénine, soit avec un fluorochrome) et de la Taq ADN polymérase est ajouté sur la lame. La première étape consiste en l’appariement des amorces non marquées à leur séquence d’ADN génomique complémentaire. La deuxième étape est une polymérisation à partir de l’extrémité 3’ du segment apparié par l’intermédiaire de la Taq ADN polymérase dont la fonction est de catalyser la réaction d’élongation. La présence du nucléotide marqué (dUTP sur la figure) permet la détection du fragment d’élongation nouvellement synthétisé. Dans le cas d’un couplage avec un haptène, la détection est faite à l’aide de différents systèmes de révélation (avidine-biotine, anticorps antidigoxigénine…). Dans le cas d’un marquage par un fluorochrome, la détection est directe. La durée de réalisation de la technique incluant la dénaturation des lames, la préparation des réactifs, les lavages et la révélation est d’environ 60 à 120 minutes. Les échantillons classiques d’exploitation du PRINS sont des préparations cytogénétiques, mais il est possible de réaliser ce type de réaction sur des coupes histologiques effectuées sous cryostat et sur des préparations de fibres d’ADN.
Tableau I
Comparaison des techniques FISH et PRINS.
Le développement de cette méthode a permis de multiplier et de diversifier ses applications [5, 6]. Elle permet l’analyse des différentes familles de séquences d’ADN répétées telles que : (1) les séquences de type satellite α particulières aux centromères, les séquences satellites I, II, III et les séquences β-satellites spécifiques des régions péricentromériques et hétérochromatiques [7] ; (2) les séquences répétées Alu dispersées et préférentiellement localisées dans les bandes R (reverse) des chromosomes [8] ; (3) l’ADN minisatellite des séquences télomériques [9]. La spécificité chromosomique des séquences α-satellites qui ont été décrites pour tous les chromosomes humains a été mise à profit pour développer un marquage centromérique spécifique de la plupart des chromosomes [10]. Ces données ont été utilisées pour définir des amorces oligonucléotidiques spécifiques à chacun des chromosomes en les choisissant dans la région présentant le plus de polymorphisme nucléotidique. Ainsi, chaque amorce porte un nucléotide spécifique d’un chromosome à son extrémité 3’. Parallèlement, des améliorations techniques ont porté sur l’automatisation de la technique PRINS et le marquage simultané de plusieurs chromosomes [11]. Dans le domaine de la biologie humaine, elle a principalement servi au diagnostic cytopathologique [12] et cytogénétique [13]. De récents développements techniques ont permis la détection in situ de séquences uniques [14-20], ouvrant ainsi de nouvelles perspectives pour cette technique.
Initialement, la technique PRINS était utilisée pour détecter un seul chromosome qui était révélé à l’aide d’un seul fluorochrome. Puis, divers protocoles PRINS polychromes (multicouleurs) ont été mis au point pour révéler simultanément plusieurs chromosomes. Le protocole le plus simple est réalisé en un seul cycle [20]. Il consiste à utiliser deux amorces oligonucléotidiques spécifiques de deux chromosomes donnés, dont l’une est marquée en 5’ avec un fluorochrome et l’autre non marquée. La couleur du fluorochrome utilisé pour l’élongation est différente de celle marquant l’amorce. La cible reconnue par cette amorce marquée aura une couleur composite provenant à la fois de l’hybridation de l’amorce et de l’élongation. Cette méthode permet ainsi de faire la discrimination entre les deux cibles. D’autres procédés permettent de détecter simultanément et spécifiquement plusieurs chromosomes avec des couleurs différentes sur la même cellule en interphase ou en division. Le marquage in situ de plusieurs chromosomes se fait de manière séquentielle, en utilisant plusieurs fluorochromes et en intercalant entre chaque réaction PRINS une phase de blocage au niveau de l’extrémité 3’ libre du fragment d’élongation synthétisé au cours de la réaction PRINS précédente. Une nouvelle approche plus simple et plus rapide a été récemment introduite par Yan et al. [21]. Ces auteurs ont démontré qu’à l’aide de trois amorces distinctes utilisées en trois réactions PRINS consécutives incorporant deux formes de dUTP (couplés à la biotine et à la digoxigénine) et de seulement deux fluorochromes (rhodamine et fluorescéine), il était possible de produire in situ trois marquages chromosomiques spécifiques révélés par trois couleurs différentes en omettant simplement l’étape de blocage en 3’ du segment d’élongation (Figure 2). L’obtention de trois couleurs distinctes (rouge, vert et jaune) est directement fonction de l’insertion des nucléotides marqués par un fluorochrome dans le fragment d’élongation produit au cours des trois réactions PRINS consécutives. Cette approche présente l’avantage d’éliminer l’étape de blocage qui rallonge la durée de la réaction globale. La succession de ces réactions peut être entièrement réalisée en l’espace de 90 minutes, diminuant le temps et le coût de la technique. En théorie, la combinaison Booléenne 2N- 1, où N représente le nombre de fluorochromes différents, peut être appliquée pour couvrir l’ensemble du génome humain. Cette nouvelle approche montre qu’il est possible de multiplier le nombre de couleurs et par conséquent le nombre de cibles autant de fois qu’on le souhaite.
Figure 2
Technique PRINS triple-couleurs.
A. Schéma de la technique PRINS triple-couleurs développée par Yan et al. [21]. Après trois réactions PRINS consécutives, les cibles des chromosomes 7, 8 et 18 sont identifiées respectivement par les couleurs jaune, rouge et vert. Les flèches en pointillés situées sous l’ADN chromosomique indiquent les sites d’extension sur chaque brin du début à la fin de chaque réaction PRINS. La méthode illustrée ici correspond au marquage séquentiel (trois réactions successives) utilisant la biotine, la digoxigénine et la biotine. La détection est réalisée avec l’avidine-fluorescéine et l’anti-digoxigénine-rhodamine qui se lient respectivement à la biotine et à la digoxigénine. Les meilleurs résultats, pour la création de la couleur jaune, sont obtenus avec un rapport 2 : 1 de fluorescence verte par rapport à la fluorescence rouge. Cela est dû aux intensités d’excitation et d’émission supérieures du fluorochrome rouge par rapport à celles du fluorochrome vert. B. Détection simultanée de trois chromosomes par réaction PRINS triple-couleurs sur une métaphase et un noyau interphasique. Les deux signaux jaunes correspondent aux centromères des chromosomes 7 (première cible) et sont facilement reconnaissables. Les deux signaux rouges correspondent aux centromères des chromosomes 8 (deuxième cible) et les deux signaux purement verts représentent les centromères des chromosomes 18 (troisième cible).
La technique PRINS et les autres technologies
PRINS combinée à la technique FISH
On peut utiliser simultanément des marquages FISH et PRINS sur une même préparation cellulaire, ce qui rend possible la détection d’un nombre important de chromosomes [22] et l’identification du chromosome sur lequel est observé un signal FISH par un signal PRINS centromérique ou un marquage Alu [20]. Dans les cas d’hémopathies malignes à caryotype complexe, la combinaison des deux techniques a souvent été très utile pour détecter des anomalies de nombre et de structure présentes dans un même clone cellulaire. En effet, l’utilisation d’une amorce spécifique du centromère du chromosome 8 en technique PRINS jumelée à l’utilisation de sondes spécifiques des gènes Abl (Abelson) et Bcr (breakpoint cluster region) en technique FISH a permis de mettre en évidence des anomalies associées au sein d’une même cellule et issues d’un même clone cellulaire (Figure 3 A) [23].
Figure 3
Exemples d’application de la technique PRINS.
A. La détection simultanée du chromosome philadelphie (les signaux jaunes caractérisés par un mélange de rouge et de vert sont indiqués par des flèches), du chromosome 9 (un des signaux verts sur chacun des noyaux) et du chromosome 22 (signal rouge sur chacun des noyaux) par technique FISH et la détection du chromosome 8 (tous les signaux verts sauf un) par la technique PRINS. Sur la photo figurent deux noyaux représentant deux clones différents. Le noyau de gauche contient un chromosome philadelphie (flèche) et une trisomie 8 alors que le noyau de droite montre deux chromosomes philadelphie (flèches) et une disomie 8. B. Métaphase partielle montrant la détection des télomères par PRINS sur des chromosomes de fibroblastes de souris. C. Détection d’une cellule foetale mâle présente dans le sang périphérique maternel (X en rouge et Y en vert), entourée de cellules maternelles (deux chromosomes X en rouge).
Self-primed in situ labeling (SPRINS)
Le SPRINS est une technique hybride associant la technique PRINS à la translation de coupure (nick translation) in situ. Cette technique permet de déceler les cassures d’ADN produites par des enzymes de restriction ou induites par des agents clastogènes endommageant l’ADN, comme les rayons ultraviolets. Le principe en est simple : on utilise l’extrémité 3’ libre des cassures bicaténaires comme amorce de la réaction. Après traitement par les enzymes de restriction HpaII et MspI, il est possible de détecter les îlots CpG et de faire l’étude de leur distribution et de leur méthylation sur les chromosomes humains. Les bandes chromosomiques G (Giemsa), riches en adénine et thymine, et R, riches en guanine et cytosine, possèdent des propriétés moléculaires et structurales différentes [24]. Les bandes R contiennent la plupart des gènes, notamment tous les gènes de ménage (housekeeping genes) et plusieurs gènes tissus spécifiques. On retrouve des ilôts CpG à l’extrémité 5’ des gènes de ménage et tissus spécifiques. Grâce à leur reconnaissance spécifique des séquences riches en GC, ces endonucléases combinées au SPRINS permettent un marquage chromosomique de type bandes R. Cette même technique a, par ailleurs, permis la détection du nombre de copies des chromosomes, l’analyse des différences de méthylation des chromosomes X et des chromosomes homologues acrocentriques [25].
PRINS et marquage en immunofluorescence
La capacité de marquer par fluorescence sur une même lame des cibles d’ADN et de protéines ouvre des possibilités d’investigation très intéressantes. La production simultanée de signaux par cytogénétique moléculaire et par immunologie ne semble pas aisée car, à notre connaissance, une seule publication fait état de résultats satisfaisants en utilisant la technique FISH [20]. Dans le but d’étudier la ségrégation des chromosomes durant le processus de polyploïdisation des mégacaryocytes destinés à fournir des plaquettes, nous avons, après quelques essais infructueux avec FISH, développé une méthode combinant la réaction PRINS et l’immunofluorescence. Après déshydratation, la réaction PRINS, qui permet le marquage du centromère d’un chromosome présélectionné, est immédiatement suivie par les réactions antigéniques permettant de révéler les fuseaux achromatiques via un anticorps antitubuline [26] (Figure 4). Une asymétrie dans la ségrégation des chromosomes a ainsi pu être mise en évidence [27]. Nous pensons que les avantages du PRINS tiennent, d’une part, à sa rapidité d’exécution, qui évite le contact prolongé des protéines avec la formamide contenue dans les solutions d’hybridation de la technique FISH et, d’autre part, à la masse stérique plus réduite de ses réactifs, qui assure une pénétration plus facile dans les échantillons.
Figure 4
Technique PRINS associée à l’immunofluorescence.
Exemple d’une ségrégation asymétrique des chromosomes 1 au stade anaphase des endomitoses mégacaryocytaires (4n → 8n). A. Les signaux verts (FITC : fluorescéine isothiocyanate) correspondent aux centromères des chromosomes 1. B. Les microtubules du fuseau mitotique sont immunocolorés en rouge (TRITC : tétraméthyl rhodamine isothiocyanate). C. L’ADN des noyaux est contre-coloré en bleu (iodure de TOTO3). D. Superposition des trois marqueurs (photographie reproduite avec la permission de Blood [26]).
Principales applications de la technique PRINS
Applications fondamentales de la technique PRINS
Analyse de la structure chromosomique
L’utilisation de la méthode PRINS avec des amorces spécifiques des centromères humains et des séquences Alu a permis la caractérisation d’hybrides somatiques et de lignées cellulaires tumorales. Les séquences Alu sont particulièrement concentrées au niveau des bandes R. Cette spécificité a été mise à profit pour effectuer la détection simple et rapide du matériel chromosomique humain présent dans des hybrides somatiques interespèces [8].
La technique PRINS a aussi été très largement utilisée pour localiser les séquences télomériques (TTAGGG)n sur des préparations chromosomiques d’origines diverses. Chez l’homme, cette approche a permis d’étudier les variations de la taille des séquences télomériques [28]. Un travail similaire a été réalisé dans nos laboratoires sur des fibroblastes d’embryons de souris [29] (Figure 3B) et diverses espèces d’oiseaux [20]. Chez les primates prosimiens (lémuriens), l’étude des régions télomériques a contribué à l’identification des remaniements chromosomiques à l’origine de la formation des chromosomes métacentriques et submétacentriques [30].
La colocalisation par PRINS des séquences télomériques et centromériques sur des cellules soumises à des agents bloquant la cytokinèse a aidé à comprendre les mécanismes à l’origine des micronoyaux spontanés et induits [31]. On admet que les micronoyaux peuvent résulter soit d’une malségrégation des chromosomes avec centromères et télomères présents, soit de cassures chromosomiques avec perte de centromères. L’étude de la mécanique chromosomique montre que l’accident le plus fréquent qui conduit à la formation d’un micronoyau est représenté par une non-disjonction suivie d’une perte de chromosomes. Russo et al. [32] ont décrit un protocole PRINS démontrant une double détection des séquences d’ADN satellite centromérique et péricentromérique. Cette approche a permis d’évaluer les séquences préférentiellement impliquées dans la formation des dommages chromosomiques à l’origine des micronoyaux.
Une autre application intéressante de la technique PRINS est son utilisation sur des fibres de chromatine. Le traitement des cellules avec une forte concentration de sel ou avec des détergents hyperalcalins permet de libérer la chromatine des noyaux. La chromatine décondensée (0,34 μm/kb) apparaît sous forme de fibres ou de halos d’ADN. Après révélation, les signaux fluorescents s’alignent sur la fibre à la manière d’un « collier de perles ». Cette méthode permet une mesure relativement précise de la distance physique (1 à 5 kb) entre deux séquences d’ADN connues [33].
Détection des séquences uniques d’ADN
En 1993, une étude rapportait la localisation du gène codant pour le facteur IX antihémophilique sur le chromosome X humain [34] et, en 1994, une autre étude rapportait celle de microsatellites porcins [35] par une méthode connue sous l’appellation de DISC-PCR (direct in situ single copy-PCR). Toutefois, les protocoles utilisés n’ont jamais permis de reproduire les résultats publiés. Ces échecs semblent essentiellement dus à la faible extension de l’élongation, qui ne dépasse pas une centaine de paires de bases [36].
Une étape importante a été franchie avec la publication de l’identification in situ des gènes SRY et SOX3 [14, 15]. Les protocoles développés par Kadandale et al. [15, 16] renouent avec la simplicité initiale de la technique PRINS où chaque étape a été optimisée pour amplifier fortement le signal sans produire de bruit de fond. Ces protocoles permettent une localisation efficace et reproductible de séquences uniques, sans alourdissements ou modifications considérables de la méthode de base. Outre l’utilisation de plusieurs amorces en une seule étape d’appariement et d’extension, un anticorps inactivant la Taq ADN polymérase (principe de la technique hotstart) est aussi utilisé pour augmenter la sensibilité lors de la réaction d’élongation. Cependant, l’élément novateur est un système d’amplification du signal fondé sur la technique CARD (catalyzed reporter deposition) qui utilise un composé phénolique, la tyramine biotinylée (tyramide). Cette dernière réagit rapidement avec des espèces réactives de l’oxygène au niveau du site de liaison de l’anticorps antihaptène conjugué à la peroxydase et du nucléotide marqué. La réaction, en présence d’espèces réactives de l’oxygène dérivées du peroxyde d’hydrogène (H2O2) et de tyramines couplées à un haptène non fluorescent ou à un fluorochrome, réalise le dépôt in situ de la tyramine activée. La détection du signal ainsi amplifié (jusqu’à 100 fois) se fait par méthode immunohistochimique.
Détection des séquences uniques d’ARN
Il est aussi possible de détecter les acides ribonucléiques (ARN) in situ grâce à une modification de la technique PRINS. La transcriptase inverse est utilisée pour catalyser la réaction de polymérisation de l’ADN complémentaire à l’aide de désoxyribonucléotides [37]. Cette réaction PRINS-ARN permet la détection des ARN dans les cellules hématopoïétiques en suspension, avec quantification en cytométrie de flux [38].
Applications médicales de la technique PRINS
Diagnostic prénatal invasif et non invasif
Plusieurs équipes ont rapporté l’identification d’aneuploïdies sur des prélèvements de liquide amniotique et de villosités choriales à l’aide d’amorces centromériques spécifiques, notamment celles des chromosomes 13, 16, 18, 21, X et Y [13]. Dans tous les cas étudiés, les résultats obtenus concordaient avec ceux du caryotype. La rapidité d’exécution du PRINS trouve tout son intérêt dans ce type d’application. De nouvelles approches non invasives de détection des cellules foetales présentes dans le sang maternel ont bénéficié de la technique PRINS. L’étude réalisée par Orsetti et al. [39] a permis d’identifier des cellules foetales en utilisant un protocole d’isolement par centrifugation sur double gradient d’Histopaque combiné au PRINS. Notre récente étude [40] a confirmé l’efficacité de la technique PRINS comme alternative à la technique FISH pour la quantification du nombre total de cellules foetales nucléées circulantes dans le sang maternel pendant le second trimestre de la grossesse. Un nombre total de deux à six cellules foetales par millilitre de sang a pu ainsi être déterminé (Figure 3C). Un travail similaire réalisé sur des femmes enceintes de foetus aneuploïdes est actuellement en cours. Les résultats préliminaires observés chez 10 de ces femmes montrent que le nombre de cellules foetales est en moyenne 5 fois plus élevé que dans les grossesses normales (K. Krabchi et al., données non publiées).
Études gamétiques et diagnostic préimplantatoire
Des études sur gamètes permettant l’estimation directe des taux d’aneuploïdie et de diploïdie dans les spermatozoïdes humains de sujets normaux ou porteurs de remaniements chromosomiques ont été rapportées [41]. Compte tenu de l’extrême compactage du noyau spermatique, la petite taille des amorces confère un réel avantage à la technique PRINS. L’analyse par PRINS polychrome a permis l’étude de la ségrégation méiotique de translocations réciproques sur spermatozoïdes. Le nombre important de spermatozoïdes pouvant être analysés à partir d’un seul éjaculat permet de définir avec précision l’incidence des différentes formes de déséquilibres chromosomiques présentes dans le sperme de chaque porteur de translocation [42]. L’information fournie peut s’avérer capitale lors du conseil génétique en vue d’une procréation médicalement assistée.
Une application diagnostique directe du PRINS a été testée avec succès sur des cellules blastomériques humaines [43]. Dans le cadre d’un diagnostic chromosomique préimplantatoire, la rapidité et la spécificité de la réaction PRINS constituent des paramètres importants au regard de la fenêtre d’intervention très étroite imposée par les protocoles de fécondation in vitro humaine. Enfin, l’utilisation du PRINS a récemment été étendue à l’étude chromosomique du premier globule polaire à la suite d’échecs de fécondation in vitro humaine [44].
Études oncohématologiques
La technique PRINS n’a été appliquée que récemment dans le diagnostic oncologique. Elle permet l’estimation précise du pourcentage de cellules aneuploïdes en évaluant non seulement les cellules en division, mais aussi les cellules en interphase. Dans certaines situations, la culture cellulaire ne serait donc plus obligatoire. On élimine ainsi la possibilité d’une évaluation cytogénétique erronée par l’avantage prolifératif d’un clone cellulaire génétiquement anormal. Dans cette optique, un travail réalisé dans notre laboratoire a permis d’analyser l’implication des anomalies cytogénétiques de nombre des chromosomes 7 et 8 chez 54 individus souffrant de désordres hématologiques [45]. Cette étude a clairement démontré que la trisomie 8 conférait un avantage prolifératif. Une autre étude comparative entre les techniques FISH, PRINS et cytogénétiques conventionnelles a été réalisée par G.V. Velagaleti et al. sur des cultures de moelle osseuse [46]. Par ailleurs, F. Pellestor et al. ont étudié l’hétérogénéité et l’instabilité chromosomique de plusieurs lignées tumorales coliques [47]. Enfin, B. Marie et al. ont rapporté la détection des chromosomes X et Y sur des préparations de leucocytes sanguins et médullaires et ont démontré l’intérêt de la technique PRINS dans le suivi des patients ayant fait l’objet d’une allogreffe de moelle osseuse avec un donneur de sexe opposé [48].
Détection du génome viral
Cette application très récente découle de l’adaptation de la technique PRINS aux échantillons de coupes histologiques [12, 20, 49]. Les techniques FISH et PRINS ont été utilisées avec succès pour détecter le génome viral du virus HPV (human papillomavirus) [50]. Cette étude rapporte une plus grande proportion de cellules positives par PRINS que par FISH. La meilleure sensibilité du PRINS est probablement due à la plus grande pénétrabilité de l’amorce. D’autre part, la réaction PRINS a permis la quantification des virus recombinants en déterminant le titre rétroviral et l’efficacité de la transduction virale. Cette évaluation représente une étape cruciale dans le cadre de la thérapie génique avec vecteurs viraux [51].
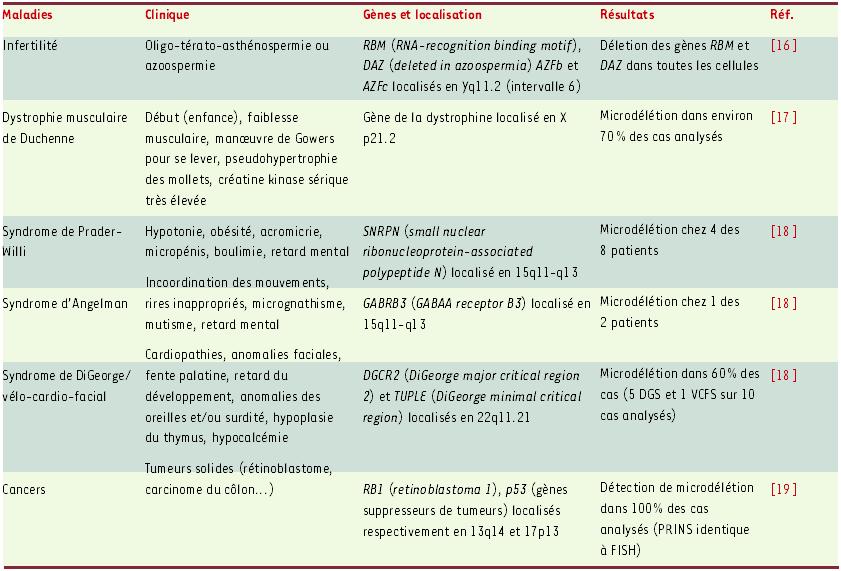
Détection des séquences uniques
Dans un numéro de l’ American Journal of Medical Genetics a été publiée une série d’articles concernant l’utilisation de la technique PRINS dans la détection de séquences uniques impliquées dans des syndromes avec microdélétion à l’échelle d’un gène. [16-19] (Tableau II).
Tableau II
Exemples d’applications de la technique PRINS dans la détection des séquences uniques d’ADN.
Concordance totale entre les résultats obtenus par PRINS et par FISH (les deux techniques ont été utilisées sur 20 métaphases et 80 noyaux interphasiques).
Il est intéressant d’évoquer certaines perspectives envisageables dans le futur, en particulier l’application de la technique PRINS dans la détection des gènes de fusion résultant d’une translocation réciproque : gène de fusion bcr/abl de la leucémie myéloïde chronique en rapport avec une translocation t(9 ; 22)(q34.1 ; q11.2) ; gène de fusion PML/RARA des leucémies aiguës myéloblastiques dû à la translocation t(15 ; 17)(q22 ; q21) ; gène de fusion EWS/FLI-1, observé dans 85 % des sarcomes d’Ewing, en rapport avec une translocation t(11 ; 22)(q24 ; q12).
Conclusions
Actuellement, la réaction PRINS peut être très avantageusement combinée à la technique FISH dans les cas où un gène de fusion et une aneuploïdie doivent être recherchés dans une même cellule. La technique PRINS devrait remplacer la technique FISH dans toutes les situations de détermination du sexe chromosomique, de recherche d’aneuploïdie et de diagnostic d’un syndrome microdélétionnel.
Par les nombreux avantages qu’elle offre, la technique PRINS constitue un outil puissant, simple et universel adapté à la cytogénétique moléculaire. Elle représente une excellente alternative à la technique FISH tant sur le plan de la recherche scientifique que de l’investigation génétique clinique en pratique médicale. Son développement et sa diffusion dans les laboratoires de cytogénétique témoignent de son efficacité. Les progrès techniques qui en découlent renforcent les espérances fondées sur cette technique. La détection in situ de séquences uniques apporte une contribution significative à l’avancement des connaissances en biologie appliquée particulièrement dans la compréhension de certaines entités nosologiques. Cet apport constitue une réelle consécration de la technique PRINS et contribue largement à en faire une technique de choix de cytogénétique moléculaire.
Parties annexes
Remerciements
Les travaux présentés dans cet article ont été financés par le Réseau Canadien des Maladies Génétiques (CGDN) et le Réseau de Médecine Génétique Appliquée (RMGA) du Québec. Les travaux de P. Coullin ont bénéficié de fonds provenant de l’Inserm et du CNRS. La Figure 4 a été réalisée au cours de travaux faits en collaboration avec Lydia Roy (Inserm U.362, Villejuif). Les travaux de F. Pellestor sur le PRINS sont financés par une subvention de l’Association Française contre les Myopathies (AFM) et un contrat européen COPERNICUS-2 (ICA2-CT2000-10012). R. Drouin détient une bourse de chercheur-boursier (niveau senior) du Fonds de la Recherche en Santé du Québec (FRSQ).
Références
- 1. Pardue ML, Gall JG. Molecular hybridization of radioactive DNAto the DNA of cytological preparations. Proc Natl Acad Sci USA 1969 ; 64 : 600-4.
- 2. Pinkel D, Straume T, Gray JW. Cytogenetic analysis using quantitative, high-sensitivity, fluorescence hybridization. Proc Natl Acad Sci USA 1986 ; 83 : 2934-8.
- 3. Cremer T, Landegent J, Bruckner A, et al. Detection of chromosome aberrations in the human interphase nucleus by visualization of specific target DNAs with radioactive and non-radioactive in situ hybridization techniques : diagnosis of trisomy 18 with probe L1.84. Hum Genet 1986 ; 74 : 346-52.
- 4. Koch JE, Kolvraa S, Petersen KB, et al. Oligonucleotide-priming methods for the chromosome-specific labelling of alpha satellite DNA in situ. Chromosoma 1989 ; 98 : 259-65.
- 5. Pellestor F. Ce que le PRINS peut faire pour vous. Med Sci 1998 ; 14 : 935-8.
- 6. Bouayed Abdelmoula N, Portnoï MF, Vialard F, et al. Les techniques de cytogénétique moléculaire : principes et progrès. Med Sci 2000 ; 16 : 1405-11.
- 7. Therkelsen AJ, Nielsen A, Kolvraa S. Localisation of the classical DNA satellites on human chromosomes as determined by primed in situ labelling (PRINS). Hum Genet 1997 ; 100 : 322-6.
- 8. Coullin P, Andreo B, Charlieu JP, et al. Primed in situ (PRINS) labelling with Alu and satellite primers for rapid characterization of human chromosomes in hybrid cell lines. Chromosome Res 1997 ; 5 : 307-12.
- 9. Therkelsen AJ, Nielsen A, Koch J, et al. Staining of human telomeres with primed in situ labeling (PRINS). Cytogenet Cell Genet 1995 ; 68 : 115-8.
- 10. Koch J, Hindkjaer J, Kolvraa S, Bolund L. Construction of a panel of chromosome-specific oligonucleotide probes (PRINS-primers) useful for the identification of individual human chromosomes in situ. Cytogenet Cell Genet 1995 ; 71 : 142-7.
- 11. Speel EJ, Lawson D, Hopman AH, Gosden J. Multi-PRINS : multiple sequential oligonucleotide primed in situ DNA synthesis reactions label specific chromosomes and produce bands. Hum Genet 1995 ; 95 : 29-33.
- 12. Wilkens L, Tchinda J, Komminoth P, Werner M. Single- and double-color oligonucleotide primed in situ labeling (PRINS): applications in pathology. Histochem Cell Biol 1997 ; 108 : 439-46.
- 13. Pellestor F, Girardet A, Lefort G, et al. Use of the primed in situ labelling (PRINS) technique for a rapid detection of chromosomes 13, 16, 18, 21, X and Y. Hum Genet 1995 ; 95 : 12-7.
- 14. Kadandale JS, Wachtel SS, Tunca Y, et al. Localization of SRY by primed in situ labeling in XX and XY sex reversal. Am J Med Genet 2000 ; 95 : 71-4.
- 15. Kadandale JS, Tunca Y, Tharapel AT. Chromosomal localization of single copy genes SRY and SOX3 by primed in situ labeling (PRINS). Microb Comp Genomics 2000 ; 5 : 71-4.
- 16. Kadandale JS, Wachtel SS, Tunca Y, et al. Deletion of RBM and DAZ in azoospermia : evaluation by PRINS. Am J Med Genet 2002 ; 107 : 105-8.
- 17. Cinti C, Stuppia L, Maraldi NM. Combined use of PRINS and FISH in the study of the dystrophin gene. Am J Med Genet 2002 ; 107 : 115-8.
- 18. Tharapel AT, Kadandale JS, Martens PR, et al. Prader Willi/Angelman and DiGeorge/Velocardiofacial syndrome deletions : diagnosis by primed in situ labeling (PRINS). Am J Med Genet 2002 ; 107 : 119-22.
- 19. Tharapel SA, Kadandale JS. Primed in situ labeling (PRINS) for evaluation of gene deletions in cancer. Am J Med Genet 2002 ; 107 : 123-6.
- 20. Coullin P, Roy L, Pellestor F, et al. PRINS, the other in situ labeling method useful in cellular biology. Am J Med Genet 2002 ; 107 : 127-35.
- 21. Yan J, Bronsard M, Drouin R. Creating a new color by omission of 3 end blocking step for simultaneous detection of different chromosomes in multi-PRINS technique. Chromosoma 2001 ; 109 : 565-70.
- 22. Pellestor F, Quenesson I, Coignet L, et al. FISH and PRINS, a strategy for rapid chromosome screening : application to the assessment of aneuploidy in human sperm. Cytogenet Cell Genet 1996 ; 72 : 34-6.
- 23. Yan J, Marceau D, Drouin R. Tetrasomy 8 is associated with a major cellular proliferative advantage and a poor prognosis. Two cases of myeloid hematologic disorders and review of the literature. Cancer Genet Cytogenet 2001 ; 125 : 14-20.
- 24. Drouin R, Holmquist GP, Richer CL. High-resolution replication bands compared with morphologic G- and R-bands. Adv Hum Genet 1994 ; 22 : 47-115.
- 25. Andersen CL, Koch J, Kjeldsen E. CpG islands detected by self-primed in situ labeling (SPRINS). Chromosoma 1998 ; 107 : 260-6.
- 26. Roy L, Coullin P, Vitrat N, et al. Asymmetrical segregation of chromosomes with a normal metaphase/anaphase checkpoint in polyploid megakaryocytes. Blood 2001 ; 97 : 2238-47.
- 27. Saffery R, Irvine DV, Griffiths B, et al. Human centromeres and neocentromeres show identical distribution patterns of > 20 functionally important kinetochore-associated proteins. Hum Mol Genet 2000 ; 9 : 175-85.
- 28. Krejci K, Koch J. An in situ study of variant telomeric repeats in human chromosomes. Genomics 1999 ; 58 : 202-6.
- 29. Lavoie J, Bronsard M, Lebel M, Drouin R. Mouse telomere analysis using an optimized primed in situ (PRINS) labeling technique. Chromosoma 2003 ; 111 : 438-44.
- 30. Go Y, Rakotoarisoa G, Kawamoto Y, et al. PRINS analysis of the telomeric sequence in seven lemurs. Chromosome Res 2000 ; 8 : 57-65.
- 31. Russo A, Priante G, Tommasi AM. PRINS localization of centromeres and telomeres in micronuclei indicates that in mouse splenocytes chromatid non-disjunction is a major mechanism of aneuploidy. Mutat Res 1996 ; 372 : 173-80.
- 32. Russo A. PRINS tandem labeling of satellite DNA in the study of chromosome damage. Am J Med Genet 2002 ; 107 : 99-104.
- 33. Shibasaki Y, Gosden JR. Oligonucleotide PRINS DNA synthesis on extended chromatin preparations. Methods Mol Biol 1997 ; 71 : 45-51.
- 34. Cinti C, Santi S, Maraldi NM. Localization of single copy gene by PRINS technique. Nucleic Acids Res 1993 ; 21 : 5799-800.
- 35. Troyer DL, Goad DW, Xie H, et al. Use of direct in situ single-copy (DISC) PCR to physically map five porcine microsatellites. Cytogenet Cell Genet 1994 ; 67 : 199-204.
- 36. Coullin P, Valent A, Barbounaki I, et al. Rapid identification of chromosomes by in situ hybridization of labelled oligonucleotides and comparison with the PRINS method. CR Acad Sci III 1996 ; 319 : 901-6.
- 37. Mogensen J, Kolvraa S, Hindkjaer J, et al. Nonradioactive, sequence-specific detection of RNA in situ by primed in situ labeling (PRINS). Exp Cell Res 1991 ; 196 : 92-8.
- 38. Bains MA, Agarwal R, Pringle JH, et al. Flow cytometric quantitation of sequence-specific mRNA in hemopoietic cell suspensions by primer-induced in situ (PRINS) fluorescent nucleotide labeling. Exp Cell Res 1993 ; 208 : 321-6.
- 39. Orsetti B, Lefort G, Boulot P, et al. Fetal cells in maternal blood : the use of primed in situ (PRINS) labelling technique for fetal cell detection and sex assessment. Prenat Diagn 1998 ; 18 : 1014-22.
- 40. Krabchi K, Gros-Louis F, Yan J, et al. Quantification of all fetal nucleated cells in maternal blood between the 18th and 22nd weeks of pregnancy using molecular cytogenetic techniques. Clin Genet 2001 ; 60 : 145-50.
- 41. Pellestor F, Girardet A, Coignet L, et al. Assessment of aneuploidy for chromosomes 8, 9, 13, 16, and 21 in human sperm by using primed in situ labeling technique. Am J Hum Genet 1996 ; 58 : 797-802.
- 42. Pellestor F, Girardet A, Coignet L, et al. Cytogenetic analysis of meiotic segregation in sperm from two males heterozygous for reciprocal translocations using PRINS and humster techniques. Cytogenet Cell Genet 1997 ; 78 : 202-8.
- 43. Pellestor F, Girardet A, Andreo B, et al. The PRINS technique : potential use for rapid preimplantation embryo chromosome screening. Mol Hum Reprod 1996 ; 2 : 135-8.
- 44. Petit C, Martel-Petit V, Fleurentin A, et al. Use of PRINS for preconception screening of polar bodies for common aneuploidies. Prenat Diagn 2000 ; 20 : 1067-71.
- 45. Yan J, Zhang XX, Fetni R, Drouin R. Trisomy 8 and monosomy 7 detected in bone marrow using primed in situ labeling, fluorescence in situ hybridization, and conventional cytogenetic analyses. A study of 54 cases with hematological disorders. Cancer Genet Cytogenet 2001 ; 125 : 30-40.
- 46. Velagaleti GV, Tharapel SA, Tharapel AT. Validation of primed in situ labeling (PRINS) for interphase analysis : comparative studies with conventional fluorescence in situ hybridization and chromosome analyses. Cancer Genet Cytogenet 1999 ; 108 : 100-6.
- 47. Pellestor F, Andreo B, Coullin P. Interphasic analysis of aneuploidy in cancer cell lines using primed in situ labeling. Cancer Genet Cytogenet 1999 ; 111 : 111-8.
- 48. Marie B, Antunes L, Baylac F, et al. The PRINS technique. Application to the detection of X and Y chromosomes for follow-up of bone marrow allografts performed with an opposite sex donor. Ann Pathol 1997 ; 17 : 11-6.
- 49. Speel EJ, Lawson D, Ramaekers FC, et al. PRINS DNA synthesis on frozen tissue sections. Methods Mol Biol 1997 ; 71 : 31-7.
- 50. Ramael M, Van Steelandt H, Stuyven G, et al. Detection of human papilloma virus (HPV) genomes by the primed in situ (PRINS) labelling technique. Pathol Res Pract 1999 ; 195 : 801-7.
- 51. Claudio PP, Cinti C, Giordano A. Application of the primer in situ DNA synthesis (PRINS) technique to titer recombinant virus and evaluation of the efficiency of viral transduction. Anal Biochem 2001 ; 291 : 96-101.
Liste des figures
Figure 1
Schéma illustrant les deux étapes de la technique PRINS (marquage non isotopique par synthèse in situ amorcée) et ses principales applications.
Un volume d’environ 45 μl d’un mélange réactionnel constitué entre autre d’une amorce (courte séquence oligonucléotidique de 18 à 35 nucléotides), de trois nucléotides non marqués et d’un nucléotide marqué (soit avec un haptène non fluorescent comme la biotine ou la digoxigénine, soit avec un fluorochrome) et de la Taq ADN polymérase est ajouté sur la lame. La première étape consiste en l’appariement des amorces non marquées à leur séquence d’ADN génomique complémentaire. La deuxième étape est une polymérisation à partir de l’extrémité 3’ du segment apparié par l’intermédiaire de la Taq ADN polymérase dont la fonction est de catalyser la réaction d’élongation. La présence du nucléotide marqué (dUTP sur la figure) permet la détection du fragment d’élongation nouvellement synthétisé. Dans le cas d’un couplage avec un haptène, la détection est faite à l’aide de différents systèmes de révélation (avidine-biotine, anticorps antidigoxigénine…). Dans le cas d’un marquage par un fluorochrome, la détection est directe. La durée de réalisation de la technique incluant la dénaturation des lames, la préparation des réactifs, les lavages et la révélation est d’environ 60 à 120 minutes. Les échantillons classiques d’exploitation du PRINS sont des préparations cytogénétiques, mais il est possible de réaliser ce type de réaction sur des coupes histologiques effectuées sous cryostat et sur des préparations de fibres d’ADN.
Figure 2
Technique PRINS triple-couleurs.
A. Schéma de la technique PRINS triple-couleurs développée par Yan et al. [21]. Après trois réactions PRINS consécutives, les cibles des chromosomes 7, 8 et 18 sont identifiées respectivement par les couleurs jaune, rouge et vert. Les flèches en pointillés situées sous l’ADN chromosomique indiquent les sites d’extension sur chaque brin du début à la fin de chaque réaction PRINS. La méthode illustrée ici correspond au marquage séquentiel (trois réactions successives) utilisant la biotine, la digoxigénine et la biotine. La détection est réalisée avec l’avidine-fluorescéine et l’anti-digoxigénine-rhodamine qui se lient respectivement à la biotine et à la digoxigénine. Les meilleurs résultats, pour la création de la couleur jaune, sont obtenus avec un rapport 2 : 1 de fluorescence verte par rapport à la fluorescence rouge. Cela est dû aux intensités d’excitation et d’émission supérieures du fluorochrome rouge par rapport à celles du fluorochrome vert. B. Détection simultanée de trois chromosomes par réaction PRINS triple-couleurs sur une métaphase et un noyau interphasique. Les deux signaux jaunes correspondent aux centromères des chromosomes 7 (première cible) et sont facilement reconnaissables. Les deux signaux rouges correspondent aux centromères des chromosomes 8 (deuxième cible) et les deux signaux purement verts représentent les centromères des chromosomes 18 (troisième cible).
Figure 3
Exemples d’application de la technique PRINS.
A. La détection simultanée du chromosome philadelphie (les signaux jaunes caractérisés par un mélange de rouge et de vert sont indiqués par des flèches), du chromosome 9 (un des signaux verts sur chacun des noyaux) et du chromosome 22 (signal rouge sur chacun des noyaux) par technique FISH et la détection du chromosome 8 (tous les signaux verts sauf un) par la technique PRINS. Sur la photo figurent deux noyaux représentant deux clones différents. Le noyau de gauche contient un chromosome philadelphie (flèche) et une trisomie 8 alors que le noyau de droite montre deux chromosomes philadelphie (flèches) et une disomie 8. B. Métaphase partielle montrant la détection des télomères par PRINS sur des chromosomes de fibroblastes de souris. C. Détection d’une cellule foetale mâle présente dans le sang périphérique maternel (X en rouge et Y en vert), entourée de cellules maternelles (deux chromosomes X en rouge).
Figure 4
Technique PRINS associée à l’immunofluorescence.
Exemple d’une ségrégation asymétrique des chromosomes 1 au stade anaphase des endomitoses mégacaryocytaires (4n → 8n). A. Les signaux verts (FITC : fluorescéine isothiocyanate) correspondent aux centromères des chromosomes 1. B. Les microtubules du fuseau mitotique sont immunocolorés en rouge (TRITC : tétraméthyl rhodamine isothiocyanate). C. L’ADN des noyaux est contre-coloré en bleu (iodure de TOTO3). D. Superposition des trois marqueurs (photographie reproduite avec la permission de Blood [26]).
Liste des tableaux
Tableau I
Comparaison des techniques FISH et PRINS.
Tableau II
Exemples d’applications de la technique PRINS dans la détection des séquences uniques d’ADN.