Corps de l’article
Les dystrophinopathies sont des affections liées à des anomalies de la dystrophine, protéine sub-sarcolemmique de 427 kDa. Celle-ci est soit absente (dystrophie musculaire progressive et sévère de Duchenne), soit présente mais diminuée et/ou altérée qualitativement (dystrophies moins sévères de type Becker). Cette hétérogénéité clinique est sous-tendue par une hétérogénéité des mutations dans le gène DMD (2,4 Mb, 79 exons) qui est l’objet de mutations très variables que l’on peut regrouper en deux catégories affectant différemment la synthèse de la dystrophine : (1) les grands remaniements touchant un ou plusieurs exons (délétions dans 60 %-70 % des cas, duplications dans 10 %-15 % des cas) ; (2) les « petites » mutations intra- ou juxta-exoniques (mini-insertions ou délétions, mutations ponctuelles de type non-sens direct, mutations d’épissage).
En règle générale, la forme de Duchenne est due à des mutations nulles, incompatibles avec une quelconque production de dystrophine (mutations interrompant le cadre de lecture, comme les mutations non-sens direct, les délétions ou insertions créant un codon non-sens prématuré). Les formes moins sévères de type Becker sont dues à des mutations compatibles avec la production d’une certaine quantité de dystrophine. En ce qui concerne les grandes délétions internes, de loin les plus fréquentes, la sévérité du phénotype (Duchenne ou Becker) n’est pas conditionnée par leur étendue mais par l’impact sur le cadre de lecture. De très grandes délétions peuvent n’entraîner qu’un tableau clinique peu sévère si le cadre de lecture est préservé. En effet, la distribution des 11 055 nucléotides de la séquence codante de l’isoforme musculaire de la dystrophine (Dp427 = 427 kDa) sur les 79 exons du gène DMD ne coïncide pas régulièrement avec le découpage en triplets. Ainsi, la dystrophine, avec sa structure modulaire, et notamment son domaine central constitué de 24 répétitions de motifs de type spectrine (109 acides aminés), est une protéine tolérant l’ablation de certaines régions internes, pourvu que la phase de la séquence codante finale soit préservée. Cela a conduit au concept de modèles réduits mais encore fonctionnels de la dystrophine : mini-dystrophine, amputée de 46 % de la séquence peptidique [1] et micro-dystrophine, amputée de 60 % [2], très utilisées dans les essais de thérapie génique par transfert de gène. Compte tenu de l’organisation des exons du gène DMD, on conçoit qu’une phase abolie par une mutation (délétion ou mutation ponctuelle) puisse être rétablie par l’exclusion d’un ou plusieurs exons si la phase locale s’y prête. C’est ce qu’on appelle le saut d’exon thérapeutique (SET) (en anglais, exon skipping). D’où l’idée d’intervenir sur le processus d’épissage pour empêcher l’incorporation de l’exon ou des exons choisis. Ces sauts d’exon peuvent être induits par des oligonucléotides antisens (modifiés pour résister aux RNases), complémentaires de séquences-clés qui président à l’épissage du transcrit primaire. Dès 1991, M. Matsuo [3] avait insisté sur l’importance des phénomènes d’épissage dans le déterminisme et la thérapeutique des dystrophinopathies. Plus récemment, d’autres groupes se sont engagés dans la voie de l’épissothérapie, comme alternative à la thérapie génique classique qui n’a pas encore remporté les succès escomptés [4-6]. Une restauration de la synthèse de dystrophine musculaire chez la souris mdx par exclusion post-transcriptionnelle de l’exon porteur de la mutation (stop sur l’exon 23) a été rapportée par plusieurs équipes [5, 6]. Mais ni l’efficacité, ni la stabilité n’atteignaient un niveau de significativité clinique.
Dans une publication récente [7], nous décrivons une nouvelle stratégie d’épissothérapie qui a permis d’obtenir, chez la souris mdx, une restauration efficace et durable de la synthèse de dystrophine. Un mois après une injection unique intramusculaire ou intra-artérielle d’un vecteur viral, l’AAV (adeno-associated virus), portant diverses constructions (Figure 1) destinées à empêcher l’incorporation de l’exon 23 dans le transcrit final, la quasi-totalité des cellules du muscle injecté, ou des muscles du membre perfusé, réexpriment un taux normal de dystrophine au niveau du sarcolemme (Figures 2 et 3). Parallèlement, on observe la réapparition à la membrane des protéines du complexe des glycoprotéines associées à la dystrophine, et une restauration de l’intégrité membranaire attestée par l’imperméabilité aux colorants vitaux. Enfin, la plupart des fibres musculaires ont récupéré une résistance normale à l’effort. Tous ces effets sont étonnamment stables et se sont maintenus pendant 6 mois, recul dont nous disposons aujourd’hui. Nous pensons ainsi avoir atteint un niveau de significativité clinique. Le succès de la stratégie tient à la nature des constructions employées : au lieu d’injecter des oligonucléotides antisens libres, nous avons combiné l’épissothérapie par oligonucléotide antisens à une technologie classique de thérapie génique. Nous voulions obtenir, avec une injection unique, une synthèse endogène d’antisens par un minigène, et un effet thérapeutique durable. Ici, le produit du minigène consiste en une séquence antisens appropriée, camouflée dans un petit ARN nucléaire naturel, le snARN U7, qui est normalement impliqué dans la maturation des transcrits d’histone (Figure 1) [8]. L’ARN U7 chimère est produit sous l’influence de son propre promoteur ; l’antisens antagoniste d’épissage qu’il contient n’est pas reconnu comme une séquence exogène et échappe à la dégradation par les RNases ; enfin, et surtout, il est produit en permanence. La vectorisation dans le virus AAV (sérotype 2/1) est aussi un élément très important, car ce vecteur est très efficace pour la transduction des cellules musculaires.
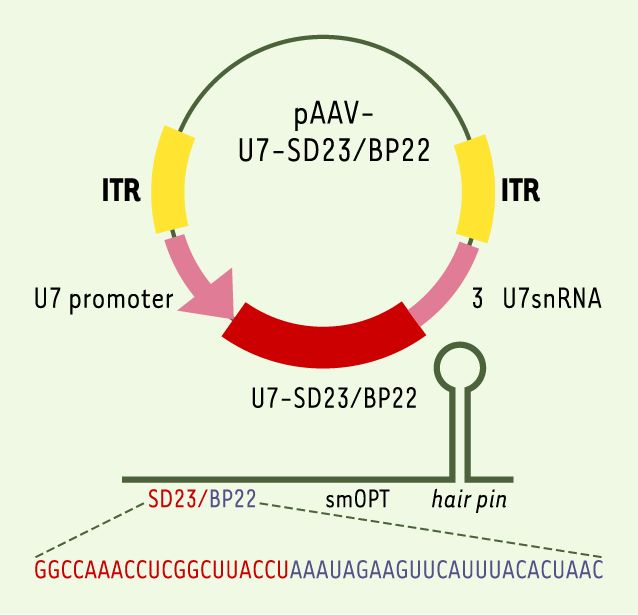
Figure 1
Construction pAAV-U7-SD23/BP22.
Le U7snARN transformé en U7-SD23/BP22 spécifique de l’exon 23 du gène de la dystrophine murine est cloné entre les deux ITR d’un vecteur AAV2, encapsidé ensuite dans un vecteur AAV de type 1. Ce U7snARN est inséré sous le contrôle de son propre promoteur et de ses régions 3’ régulatrices, permettant l’expression d’un petit ARN modifié U7-SD23/BP22.
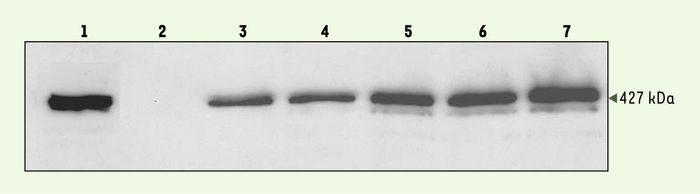
Figure 2
Western blot sur protéines totales extraites des muscles de souris mdx injectées.
Détection de la dystrophine avec l’anticorps monoclonal de souris NCL-DYS1 (Novocastra). La pointe de flèche indique la taille de la dystrophine normale (427 kDa) qui est détectée dans un muscle de souris normale C57Bl6 (échantillon 1). L’échantillon 2 représente le muscle mdx non traité, et les échantillons 3 à 7 correspondent respectivement aux muscles injectés avec l’AAV-U7(SD23/BP22), 2, 4, 8, 13 et 26 semaines après traitement. La différence de taille attendue, d’environ 8 kDa entre la dystrophine normale et la dystrophine restaurée, ne peut être détectée sur ce gel.
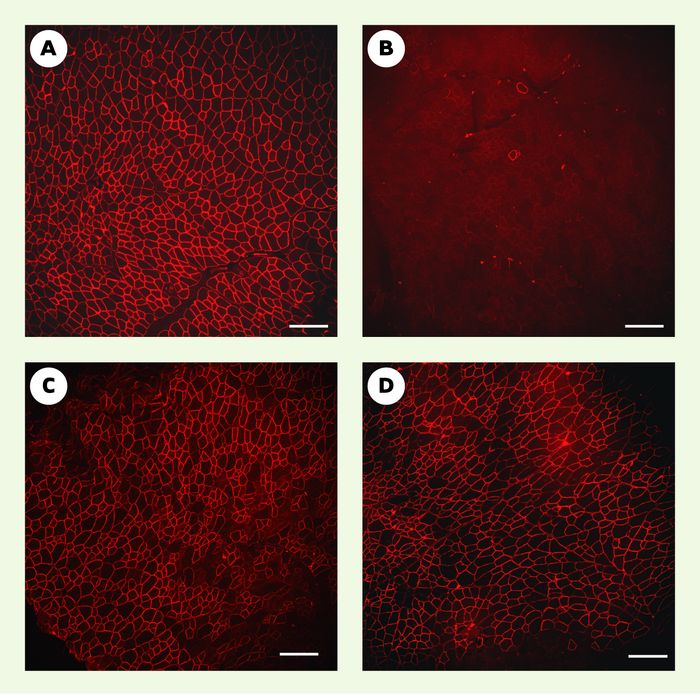
Figure 3
Immunomarquage de la dystrophine avec l’anticorps monoclonal de souris NCL-DYS2 (Novocastra).
Immunomarquage sur sections de muscles (tibialis anterior) de souris normale C57Bl6 (A), de souris mdx non traitée (B), de souris mdx 1 mois (C) et 6 mois (D) après l’injection de l’AAV-U7-SD23/BP22 (barres d’échelle [A-D] : 0,5 mm).
Ces résultats encourageants suggèrent que la stratégie utilisée pourrait être employée en thérapeutique humaine. Un premier problème concerne l’éligibilité des patients pour une telle approche. L’objectif que l’on vise par le SET est d’obtenir un taux suffisant de transcrit lisible pour aboutir à une quantité de protéine fonctionnelle capable de contrecarrer le processus pathologique. Comme la dystrophine finale est amputée d’un ou plusieurs exons, on peut prédire qu’elle se comportera comme une dystrophine de type Becker. Cette assertion est objectivement étayée sur les phénotypes Becker modérés, observés chez les malades dont la délétion est identique à celle qui serait obtenue par SET. Autrement dit, dans la majorité des cas, il s’agit d’obtenir la transformation d’un phénotype DMD très sévère, en phénotype BMD. Dans les rares cas où il existe déjà un certain taux basal de transcrit normal, on peut espérer corriger la copie anormale du transcrit et obtenir la transformation d’un phénotype BMD en phénotype normal. Grâce à la base de données UMD-DMD, développée par Christophe Béroud à partir de son modèle générique de base de mutations spécifiques de locus [9], nous disposons d’un moyen de sélectionner instantanément les cas éligibles pour un SET. En effet, cette base, qui renferme plus de 800 cas documentés au point de vue moléculaire, biologique et clinique, fournit automatiquement pour chaque cas tous les paramètres requis : (1) conséquences directes de toute mutation sur le cadre de lecture ; (2) tous profils d’épissage qui seraient correcteurs de la phase ; (3) taille de la dystrophine résultante ; (4) domaines protéiques et épitopes qui seraient sacrifiés par le SET ; (5) carte des cibles à viser par les oligonucléotides antisens pour obtenir un SET. Les mutations justiciables du SET sont les délétions génomiques, les mutations intra-exoniques bloquant le cadre de lecture et les mutations introniques réactivant des exons cryptiques. Si l’on s’en tient au saut d’un seul exon (mais le saut de plusieurs exons est envisageable), plus de 300 cas seraient éligibles, soit 43 % de l’effectif total des patients figurant dans la base UMD-DMD (83 % si l’on ne tient compte que de la population présentant une grande délétion ≥ 1 exon). Il faut néanmoins souligner qu’il s’agirait d’une thérapeutique à la carte, c’est-à-dire spécifique d’allèle. Les exons à faire sauter sont de fréquence très inégale : avec 6 exons (45, 53, 51, 44, 50 et 8), on pourrait corriger 85 % des délétions hors-phases (pour corriger 100 % des cas éligibles, il faudrait cibler en tout 22 exons). Pour la correction des petites mutations, le problème se complique par la grande diversité des exons à cibler et les très faibles effectifs.
Un autre problème à résoudre avant de passer aux essais cliniques est l’adressage. Par l’expérimentation animale (souris et chien myopathes), nous nous employons à l’heure actuelle à élaborer une méthode d’administration systémique permettant d’obtenir un adressage généralisé à tous les muscles, en particulier aux muscles respiratoires et cardiaque, et une bonne tolérance pharmacologique et immunologique. Enfin, nous soulignons qu’a priori cette stratégie n’est applicable qu’aux maladies génétiques résultant de mutations récessives, touchant des gènes codant pour des protéines modulaires contenant des motifs non essentiels. À cet égard, les dystrophinopathies apparaissent comme un cas de figure exceptionnellement privilégié.
Parties annexes
Références
- 1. England S, Nicholson L, Johnson M, et al. Very mild muscular dystrophy associated with the deletion of 46 % of dystrophin. Nature 1990 ; 343 : 180-2.
- 2. Harper SQ, Hauser MA, DelloRusso C, et al. Modular flexibility of dystrophin : implications for gene therapy of Duchenne muscular dystrophy. Nat Med 2002 ; 8 : 253-61.
- 3. Matsuo M, Masumura T, Nishio H, et al. Exon skipping during splicing of dystrophin mRNA precursor due to an intraexon deletion in the dystrophin gene of Duchenne muscular dystrophy kobe. J Clin Invest 1991 ; 87 : 2127-31.
- 4. Aartsma-Rus A, Bremmer-Bout M, Janson AA, et al. Targeted exon skipping as a potential gene correction therapy for Duchenne muscular dystrophy. Neuromuscul Disord 2002 ; 12 (suppl 1) : S71-7.
- 5. Dunckley MG, Manoharan M, Villiet P, et al. Modification of splicing in the dystrophin gene in cultured Mdx muscle cells by antisense oligoribonucleotides. Hum Mol Genet 1998 ; 7 : 1083-90.
- 6. Lu QL, Mann CJ, Lou F, et al. Functional amounts of dystrophin produced by skipping the mutated exon in the mdx dystrophic mouse. Nat Med 2003 ; 9 : 1009-14.
- 7. Goyenvalle A, Vulin A, Fougerousse F, et al. Rescue of dystrophic muscle through U7 snRNA-mediated exon skipping. Science Express Online 2004, 4 novembre, 1104297 (à paraître le 3 décembre 2004).
- 8. Gorman L, Suter D, Emerick V, et al. Stable alteration of pre-mRNA splicing patterns by modified U7 small nuclear RNAs. Proc Natl Acad Sci USA 1998 ; 95 : 4929-34.
- 9. Béroud C, Collod-Beroud G, Boileau C, et al. UMD (universal mutation database) : a generic software to build and analyze locus-specific databases. Hum Mutat 2000 ; 15 : 86-94.
Liste des figures
Figure 1
Construction pAAV-U7-SD23/BP22.
Le U7snARN transformé en U7-SD23/BP22 spécifique de l’exon 23 du gène de la dystrophine murine est cloné entre les deux ITR d’un vecteur AAV2, encapsidé ensuite dans un vecteur AAV de type 1. Ce U7snARN est inséré sous le contrôle de son propre promoteur et de ses régions 3’ régulatrices, permettant l’expression d’un petit ARN modifié U7-SD23/BP22.
Figure 2
Western blot sur protéines totales extraites des muscles de souris mdx injectées.
Détection de la dystrophine avec l’anticorps monoclonal de souris NCL-DYS1 (Novocastra). La pointe de flèche indique la taille de la dystrophine normale (427 kDa) qui est détectée dans un muscle de souris normale C57Bl6 (échantillon 1). L’échantillon 2 représente le muscle mdx non traité, et les échantillons 3 à 7 correspondent respectivement aux muscles injectés avec l’AAV-U7(SD23/BP22), 2, 4, 8, 13 et 26 semaines après traitement. La différence de taille attendue, d’environ 8 kDa entre la dystrophine normale et la dystrophine restaurée, ne peut être détectée sur ce gel.
Figure 3
Immunomarquage de la dystrophine avec l’anticorps monoclonal de souris NCL-DYS2 (Novocastra).